一、 背景
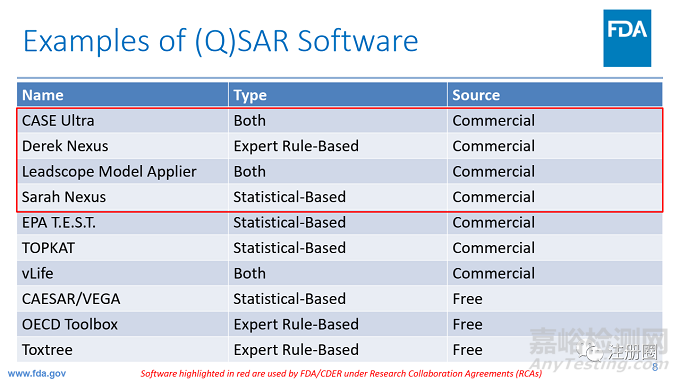
1)利用(Q)SAR軟件進行化合物的致突變性評估,無需進行復雜的毒性試驗,已越來越廣泛地應用于藥物的基因毒性雜質評估中。商業化的軟件目前市面上有好幾家,如最常用的CASE Ultra,Derek&Sarah,Leadscope等。但上述軟件的授權費用較貴,且委外評估費用也較高,基于目前雜質需要全評估的趨勢,對企業來說是一筆不小的開支。其實也有一些免費的QSAR軟件,其不僅預測準確率較高,且也被歐美法規機構所認可,不失為基因毒性雜質初步評估的一個選擇。
2)根據ICH-M7章節6中的描述:“危害性評估首先通過對數據庫和文獻的檢索獲得的雜質致癌性和細菌致突變數據對實際和潛在雜質進行初步分析,根據表 1 將其歸為 1 類, 2 類或 5 類。如果無法獲得這樣的分類數據,則應進行預測細菌致突變性的構-效關系( SAR)評估。根據評估結果將雜質歸為 3 類、 4 類或 5 類。”
指南該段話對企業而言,即應這樣進行危害性評估:先文獻和數據庫檢索雜質的相關致癌性和Ames數據,然后根據結果分類為1,2,5類;如果無法找到相關數據,應進行QSAR評估,根據評估結果分類為3類,4類和5類。
在FDA2021年3月進行了DMF培訓中,Drug Master File and Drug Substance Workshop – FDA CDER SBIA Events
在day2: ICH M7(R1) – Chemistry and Manufacturing Control (CMC) Perspective on Hazard Assessment – Barbara O. Scott 中明確指出:通過目視判斷化合物警示結構分類是不符合ICH-M7規定的。
3)在近期的各國監管評審過程中,已收到多次缺陷,需要對所有使用的物料和雜質進行致突變性評估,即使它沒有警示結果或者推測肯定是無致突性的。
Deficiency-1:QSAR or genotoxicity information for all matters
Recent trend of document examination by PMDA, QSAR for all matters is required even if they do not have alert structure.
Deficiency-2:Provide the genotoxicity data (experimental data or (Q)SAR report) of all the reagents, starting materials, intermediates and related substances involved in the manufacturing of API(MFDS)
二、 評估方法
1)采用毒性數據庫,對常見的商業化物料進行毒性數據檢索,重點進行基因毒性檢索及致癌性數據檢索。上述檢索可通過常用的一些免費數據庫來進行,也可通過免費的QSAR TOOLBOX 軟件來進行。
最常用的3個網站如下:
Lhasa Carcinogenicity Database:可查詢物質的致癌性數據及TD50值,查詢后需對結果進行判斷,判斷數據是否充分且符合ICH S1的要求。
NTP database:可查詢物質的致癌性數據及體外體內基因毒性數據,Ames實驗需判斷是否滿足OECD的指南要求,同時關注有無代謝活化情況下的結果差異。
ECHA database: 可在該物質的toxicological information中查看Genetic toxicity 和Carcinogenicity
QSAR Toolbox的數據查詢功能
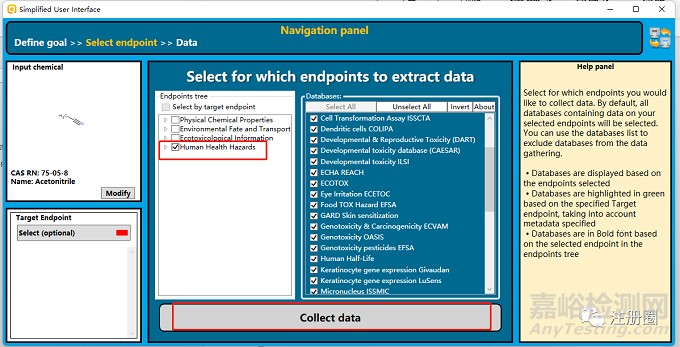
a.選用簡易用戶模式,進入結果,點擊Define輸入目標化合物
b.點擊Specific data, 勾選Human health hazards,然后點擊Collect data
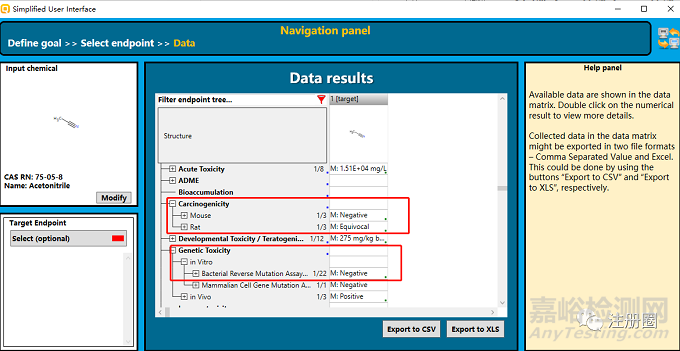
c.在data results 界面可查詢對應類別的實驗性毒性數據,重點關注遺傳毒性和致癌性,可點擊相應的數據查看數據的source 和reference。
2)采用FDA認可的免費QSAR評估軟件進行評估。FDA認可的QSAR軟件如下:
統計學規則:CAESAR/VEGA
https://www.vegahub.eu/portfolio-item/vega-qsar/
專家規則:Toxtree, OECD Toolbox,
Toxtree: https://toxtree.sourceforge.net/
OECD Toolbox: https://qsartoolbox.org/download/
對于免費QSAR軟件評估接受性的擔憂,可參考FDA 2021年3月進行了DMF培訓中說明:
CDER已經對這些免費的QSAR有了前期的知識儲備,因此可以不用再遞交經過驗證的QMRF,可直接進行結果說明即可。
Drug Master File and Drug Substance Workshop – FDA CDER SBIA Events
Application of (Q)SAR and Expert Knowledge for ICH M7 Impurity Classification– Naomi L. Kruhlak
三、 免費QSAR軟件具體使用方法:
根據ICH-M7章節6中的描述:“計算機毒理學應采用(定量)構-效關系((Q)SAR)方法學進行毒性評估,目的是預測細菌致突變試驗(參考文獻 6)的結果。應采用兩種互補的(Q)SAR 預測方法。一種方法應基于專家知識規則,另一種方法應基于統計學。(Q)SAR 模型采用的這些預測方法學應遵循經濟合作與發展組織(OECD)制訂的一般的驗證原則。如果經兩種互補的(Q)SAR 方法(專家知識規則和統計學)預測均沒有警示結構,則足以得出該雜質沒有致突變性擔憂的結論,不建議做進一步的檢測(歸為表 1 中的 5 類)。”
因此我們需要采用不同原理的兩種QSAR軟件來對雜質進行評估,具體的策略如下:
-----采用Toxtree或QSAR Toolbox 進行專家規則的評估
-----采用VEGA進行統計學規則的評估
-----若兩個評估結果都為陰性,可直接判斷為5類雜質;
如其中一個為陽性,建議對該雜質采用商業化評估軟件(如CASE Ultra,derek&sarah,leadscope)進行評估,以商業化評估軟件出具的結果為準。
3.1 Toxtree的使用方法
軟件最新版本:Toxtree 3.1.0
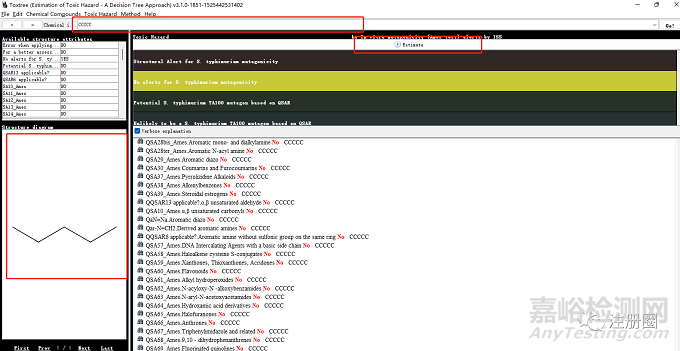
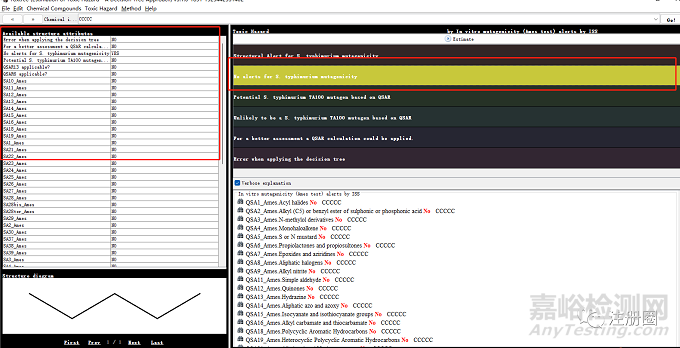
決策樹選擇:點擊最上欄中的method,點擊select a decision tree,選擇:In vitro mutagenicity (Ames test) alerts by ISS,然后點下方確定按鍵。
化合物鍵入:在Chemdraw中將化合物的結構轉化為SMILES, 復制后輸入搜索框,核對左下角的結構是否與目標結構一致,然后點擊Estimate 運行。
觀察左邊窗口的結果及中間的色條顯示,若結論為:No alerts for S.typhimurium mutagenicity,則表示為陰性。
3.2 VEGA的使用方法
軟件最新版本:
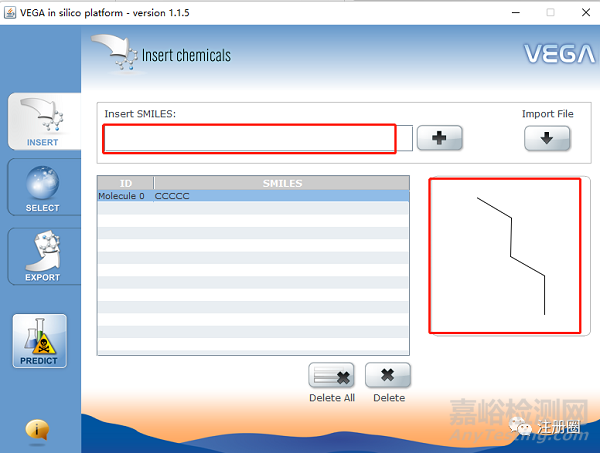
化合物鍵入:在Chemdraw中將化合物的結構轉化為SMILES, 復制后輸入搜索框,點擊+號,核對右邊顯示的結構是否與目標雜質一致。
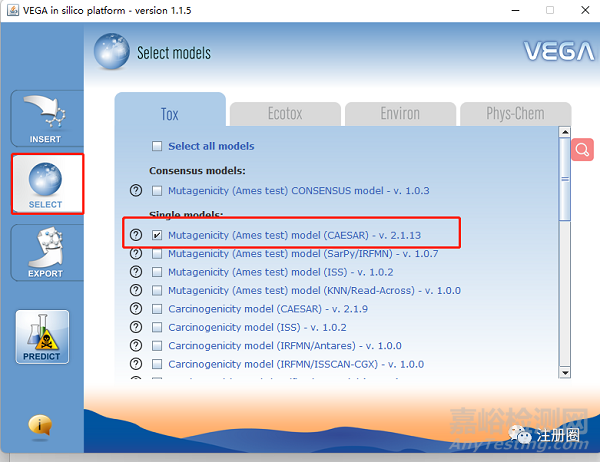
模型選擇:點擊左邊SELECT,然后選擇Mutagenicity(Ames test)model(CAESAR)-V.1.0.3
報告路徑選擇:點擊Export,選擇PDF reports(one for each model),然后選擇在計算機中報告輸出的路徑。
預測:點擊PREDICT進行預測,在設定的路徑下找到生成的預測報告,根據報告中小球的顏色及下方的評論,判斷預測的結果。綠色表示陰性,紅色代表陽性。
3.3 QSAR Toolbox的使用方法
由于QSAR Toolbox與Toxtree為同一規則,且使用的決策樹均為In vitro mutagenicity (Ames test) alerts by ISS,兩者預測結果是一樣的。考慮到Toxtree為java形式運行,使用更方便,建議直接用Toxtree即可。
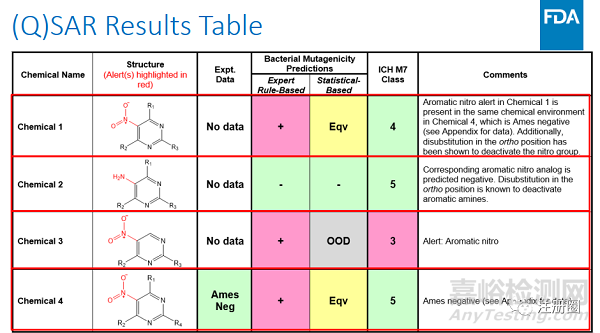
雜質評估結果在CTD資料中的書寫格式:
參照如下格式在CTD資料中提交雜質的hazard assessment summay,在comments 中加入experimental data source ,具體使用的QSAR軟件和相關的控制策略簡述。
參考文獻:
[1] ICH. Assessment and control of DNA reactive (mutagenic) impurities in pharmaceuticals to limit potential carcinogenic risk M7(R2). International Council for Harmonisation of Technical Requirements for Pharmaceuticals for Human Use (ICH)
[2] Honma M, Kitazawa A, Cayley A, et al. Improvement of quantitative structure–activity relationship (QSAR) tools for predicting Ames mutagenicity: Outcomes of the Ames/QSAR International Challenge Project. Mutagenesis. 2019; 34(1): 3– 16.
[3] Cassano, A., Raitano, G., Mombelli, E., Fernández, A., Cester, J., Roncaglioni, A., Benfenati, E., 2014. Evaluation of QSAR models for the prediction of Ames genotoxicity: a retrospective exercise on the chemical substances registered under the EU REACH regulation. J. Environ. Sci. Health C 32, 273–298.